FAQ-1032 Validierung des Dichtepunktdiagramms
density-dots-validation
Letztes Update: 16.12.2019
Dichtepunkte sind Punktdiagramme, die die Datendichte darstellen, wobei die Punkte farblich auf die Datendichte abgebildet werden. Die Dichte wird mit der Kerneldichteschätzung berechnet.
Um die Kerneldichteschätzung zu berechnen, verwendet Origin einen schnellen Algorithmus, der eine eingeteilte 2D-Approximation und 2D-Interpolation nutzt. Zuerst wird eine 2D-Einteilung für die (X-, Y-) Punkte durchgeführt, um eine Matrix mit den Klassenanzahlen zu erhalten. Dann wird die 2D-Fast-Fourier-Transformation verwendet, um diskrete Faltungen zum Berechnen der Dichtewerte von jedem Gitternetz durchzuführen. Die 4. Wurzel der Dichtewerte wird berechnet, um die Dichteskala auf die Farbskala abzubilden.
Origin kann ähnliche Ergebnisse wie R aus dem gleichen Datensatz erzeugen.
Vergleich der Ergebnisse von R und Origin
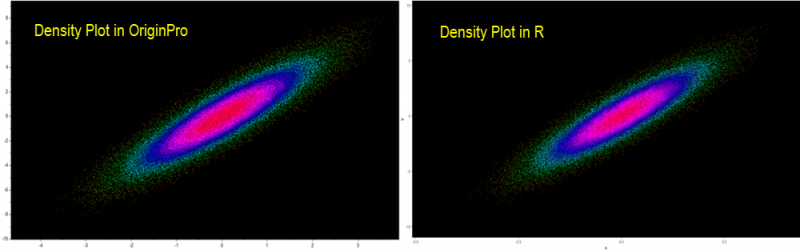
Referenz
Daten:
1 Antwortvariable (y), 1 Prädiktorvariable (x)
100000 Beobachtungen
Erzeugte Daten
Datendatei: DP.csv
Origin-Projektdatei: density_dots_validation_sample.opju
R-Codes zum Erzeugen des Diagramms
xx= read.csv("DP.csv",header=FALSE) as.list(body(smoothScatter)) trace("smoothScatter",quote(browser()),at=19) w<-c(0.056990976577602, 0.13442535086237) smoothScatter(xx, nrpoints = 0,nbin=151,bandwidth=w) #Debug and run following script #Type "n" to enter line 19 stopifnot((nx <- length(xm)) == nrow(dens), (ny <- length(ym)) == ncol(dens)) ixm <- 1L + as.integer((nx-1)*(x[,1]-xm[1])/(xm[nx]-xm[1])) iym <- 1L + as.integer((ny-1)*(x[,2]-ym[1])/(ym[ny]-ym[1])) z<-dens[cbind(ixm, iym)] df<-data.frame(x=x[,1],y=x[,2],z=z) library(ggplot2) sp<-ggplot(df, aes(x,y,colour=z)) + geom_point(shape=".") + theme(panel.background = element_rect(fill = 'black'), panel.grid.major = element_blank(), panel.grid.minor = element_blank(), panel.border = element_blank()) sp cval<-seq(0,1,length.out=6) sp+scale_color_gradientn(values = cval,colours = rainbow(20))
Schlüsselwörter:Validierung, mit R vergleichen
|